Giới thiệu
Sốt rét là một bệnh xã hội quan trọng và đe dọa sức khỏe toàn cầu, đặc biệt tại các vùng nhiệt đới và cận nhiệt đới, ước tính hàng năm có khoảng 350 - 500 triệu ca mắc mới với hơn một triệu ca tử vong do sốt rét trên toàn cẩu, tập trung phần lớn ở trẻ em nhỏ và phụ nữ mang thai. Hiện nay, bệnh đã là gánh nặng và làm ảnh hưởng hơn 2400 triệu người, trên 40% dân số thế giới, tại hơn 108 quốc gia ở vùng nhiệt đới từ Nam Mỹ đến Ấn Độ. 2/3 số ca bệnh còn lại xảy ra ở 6 nước Ấn Độ, Brazil, Sri Lanka, Việt Nam, Colombia và đảo quốc Solomon). Tổ chức Y tế thế giới (WHO) dự đoán sẽ tăng 16% số ca sốt rét mỗi năm. Khoảng chừng 1.5 – 3 triệu người chết do sốt rét mỗi năm (85% ở châu Phi). Cứ mỗi 30 giây, ở châu Phi có một trẻ chết vì sốt rét, mỗi 12 giây trên thế giới có một người chết có liên quan đến bệnh sốt rét. Mỗi năm, số người chết do sốt rét bằng số chết do HIV/AIDS trong 15 năm gộp lại. Tình hình sốt rét trong gần 10 năm qua có nhiều thay đổi một mặt do biến đổi khí hậu toàn cầu. Thời gian qua, công tác PCSR cũng đã mang lại nhiều thành quả làm giảm mắc và hạ thấp tỷ lệ sốt rét ác tính và tỷ lệ tử vong đáng kể, trong đó việc quản lý ca bệnh hiệu quả vẫn là một trong những khâu then chốt cho phòng chống sốt rét. Thành công của chiến lược này dựa trên khả năng thay đổi Chính sách thuốc sốt rét quốc gia theo từng giai đoạn nhằm cung cấp TSR có hiệu quả cao. Do vậy, đòi hỏi Chương trình phòng chống sốt rét quốc gia và các Viện chuyên ngành phải thường quy đánh giá hiệu lực TSR đang dùng và nghiên cứu thử nghiệm thuốc mới để cung cấp kịp thời và thay đổi phác đồ. Với những thông tin như thế, chúng ta có thể bảo đảm rằng quản lý ca bệnh hiệu quả thông qua việc phát hiện hoặc dự báo những mô hình kháng thuốc thay đổi sớm, từ đó thay đổi phù hợp cho Chính sách thuốc sốt rét mỗi quốc gia và thành quả của CTPCSRQG đạt được không thể không kể đến hiệu quả thuốc sốt rét.
Bệnh sốt rét đã được biết từ cổ xưa, đến nay các nhà khoa học trên thế giới tìm mọi cố gắng để đẩy lùi căn bệnh sốt rét này. Tuy nhiên, mọi cố gắng vẫn còn tiếp tục gây tốn kém nhiều trí tuệ, công sức và tiền bạc nhưng bệnh sốt rét vẫn là mối đe doạ đối với cộng đồng thế giới. Trong khi vấn đề các loài ký sinh trùng cổ điển, các nhà khoa học và cộng đồng thế giới chưa thể giải quyết triệt để, thì mới đây các nhà khoa học ở Malaysia lại công bố thêm một loài ký sinh trùng sốt rét vốn từ lâu gây bệnh sốt rét cho khỉ, nay có khả năng gây nhiễm cho người với tỷ lệ nhiễm tương đối cao và bệnh cảnh cũng có thể nghiêm trọng, vấn đề này có thể tăng thêm gánh nặng về bệnh sốt rét trong tương lai.
Việc phát hiện một loài ký sinh trùng sốt rét vốn dĩ ký sinh và gây bệnh cho người, nay lại gây bệnh và gây tử vong cho người là điều đáng quan tâm, nhân đây, bài viết nhằm tổng hợp tổ tiên và nguồn gốc của các loài ký sinh trùng sốt rét vốn dĩ trên động vật có vú liệu có khả năng lây truyền bệnh sang người hay không. Biểu đồ cây dưới đây biểu hiện liên quan giữa một số nhóm vi sinh vật, loài KSTSR đang phổ biến trên một số vật chủ hay gặp.
Phân nguồn gốc của cây phả hệ các vi sinh vật có đặc điểm trong Chi tiết của cây phả hệ di truyền hiện tại gắn liền với các vi sinh vật trong nhóm và phần còn lại của cây đời (Tree of Life). Các điểm phân nhánh từ gốc trong cây trình bày một tổ tiên của các nhóm khác trong cây. Nguồn gốc tổ tiên đa dạng tiến hóa qua thời gian đã phân thành một vài nhóm phụ (subgroup), điều này biểu hiện qua phân loại cuối cùng về phía bên phải.
Bạn có thể kích vào tên hoặc gốc các nhóm bạn quan và để chi tiết hơn, bạn có thể đi sâu vào phân cây phả hệ di truyền (phylogenetic trees) và vào phần các trang viết Phylogenetic Biology. Tài liệu Phylogeny (hay phylogenesis) dựa theo tài liệu của Escalante và cộng sự (1995); Perkins và Schall (2002); Vargas-Serrato và cộng sự (2003); Martinsen và cộng sự (2008)
Các loài ký sinh trùng sốt rét gây bệnh sốt rét ở người và loài thứ 5
Bệnh sốt rét do các ký sinh trùng (hay đơn bào đường máu) thuộc ngành Apicomplexa, giống Plasmodium gây ra. Từ trước đến nay, giới khoa học đã thừa nhận rằng bệnh sốt rét ở người chủ yếu do 4 loài P. falciparum, P. vivax, P. malariae, P. ovale gây ra, song trong số đó phần lớn là hai loài P. falciparum và P. vivax chiếm một tỷ lệ cao trong cơ cấu ký sinh trùng, tuỳ vùng địa lý mà tỷ lệ hai loại này gây nhiễm ở người có khác nhau, có nơi tỷ lệ nhiễm P. vivax đến 80% và P. falciparum chỉ 15%, có nơi P. vivax chiếm 20 – 25%, trong đó quan trọng hơn cả là loài KSTSR loại P. falciparum gây tỷ lệ sốt rét ác tính (SRAT) và tử vong cao, có nơi đến 90%. Hai loài ký sinh trùng còn lại tồn tại cũng có tỷ lệ thay đổi tuỳ theo vùng trên các châu lục.
Người ta từng được biết một số tác nhân gây bệnh mới trổi dậy (hoặc bệnh đang nổi – emerging diseases) đã lây truyền từ động vật sang người và ngược lại (zoonose). Các quần thể loài linh trưởng hoang đã có ổ chứa tiềm năng lưu giữ và là nguồn gốc của một số tác nhân gây bệnh cho người, từ các virus đến các loài giun, sán. Có hơn 26 loài Plasmodium lưu hành trong các quần thể linh trưởng hoang dã. Nhiều trong số các loài ký sinh trùng sốt rét ở loài khỉ liên quan gần các loài KSTSR gây bệnh ở người và một số trong chúng được biết là Plasmodium simium, P. brazilianum, P. cynomolgi, P. inui và P. knowlesi từng liên quan sốt rét gần biểu hiện triệu chứng ở người trong thực nghiệm hoặc nhiễm tự nhiên. Từ đầu những năm 1930, P. knowlesi đã được sử dụng để điều trị sốt trên những bệnh nhân bị giang mai thần kinh; năm 1932, Knowles và Das Gupta đã thành công trong nghiên cứu truyền bệnh sốt rét của loài khỉ cho người; năm 1960, Eyles và cộng sự lần đầu đã gây nhiễm thực nghiệm KSTSR của loài khỉ cho người thông qua gây nhiễm bằng muỗi truyền bệnh loại Plasmodium cynomolgi; năm 1967, Chin và cộng sự cho biết rằng P. knowlesi cũng có thể lây truyền từ loài khỉ qua người. Mới đây có một số báo cáo rải rác có người mắc bệnh sốt rét do P. knowlesi.
Tiếp sau Plasmodium knowlesi, liệu còn có loài KSTSR “mới” nào gây bệnh ở người?
Ca bệnh đầu tiên ở người do nhiễm tự nhiên xảy ra năm 1965 ở khu rừng rậm Malaysia, ca bệnh thứ 2 xảy ra vào năm 1971 cũng ở rừng Malaysia. Một trường hợp khác xảy ra Thái Lan (2000) ở một đàn ông 38 tuổi khi vào rừng ở biên giới Thái Lan - Myanmar. Các ước tính về tỷ lệ hiện mắc và phân bố các loài KSTSR ở các cộng đồng người nhiễm tại Malaysia đều nhờ vào chẩn đoán qua soi kính hiển vi (cacs lam máu nhuộm giêm sa) từ các phòng khám và bệnh viện nhà nước. Giữa năm 1998 và 2002, số bệnh nhân mới mắc sốt rét hàng năm ở Sarawak, thuộc quần đảo Borneo, Malaysia là 2496 - 3155 trường hợp. Ở Sarawak, loài P.vivax là loài chủ yếu (69.1%), tiếp đến P. falciparum (19.7%), rồi đến P. malariae (9.4%) và nhiễm phối hợp (# 1.0%), chưa phát hiện và khẳng định trường hợp nào do P. ovale.
Trước nghiên cứu của Singh B. và cộng sự bắt đầu vào tháng 3.2000, các nhà khoa học đã ghi nhận các khác biệt chính về các tỷ lệ của các loài Plasmodium được báo cáo ở Sarawak. P. vivax phân bố rộng rãi trong 9 vùng địa lý, trong khi P. falciparum lại chiếm ưu thế ở các vùng phía bắc của bang và KSTSR được phát hiện gần giống như P. malariae được báo cáo chủ yếu ở các vùng trung tâm của Kapit và Miri. Tại Kapit, KSTSR được phát hiện bằng kính hiển vi giống như P. malariae chiếm tỷ lệ khoảng 1/5 số trường hợp sốt rét năm 1999. Về hình thái học, nhiều lam máu có hình thái nhiễm KSTSR được phát hiện bằng kính hiển vi giống như P. malariae không điển hình. Thường là nhiễm P. malariae mãn tính và không triệu chứng với mật độ KSTSR trong máu thấp, hiếm khi vượt quá 5.000 KSTSR/μL. Tuy nhiên, hầu hết (97.4%) của 108 ca báo cáo năm 1999 ở vùng Kapit là những bệnh nhân biểu hiện đầy đủ các dấu hiệu và triệu chứng lâm sàng được nhập viện các phòng khám bệnh ở miền núi, bệnh viện Kapit và bệnh viện đa khoa Kapit. Ngoài ra, các bệnh nhân này có mật độ KSTSR trong máu 48.000 – 66.640 KSTSR/μL, với 20 ca (18.5%) có mật độ KSTSR trong máu > 5.000 KSTSR/μL.
Năm 1999, Singh B và cộng sự nghiên cứu các trường hợp sốt rét ở khu vực Kapit thuộc đảo Borneo, Malaysia qua phát hiện KSTSR bằng kính hiển vi cho thấy đó là loài P. malariae, mặc dầu hình thái xuất hiện không điển hình và họ đã dùng kỹ thuật Nested PCR để xác định P. malariae DNA nhưng không phù hợp với kết quả, họ đã tiếp tục nghiên cứu để xem rằng các trường hợp nhiễm này có phải do một biến thể của P. malariae hay do một loài Plasmodium mới xuất hiện. Để khẳng định điều đó, từ tháng 3.2000 đến 11.2002 nhóm nghiên cứu đã lấy 208 mẫu máu bệnh nhân mắc sốt rét ở khu vực Kapit để xét nghiệm so sánh song song bằng soi kính hiển vi và Nested PCR, qua phương pháp Nested PCR họ đã phát hiện 120/208 (58%) là do loài KSTSR loại P. knowlesi mà không phải do P. malariae. Triệu chứng sốt rét ở người lớn từng được báo cáo do P. malariae ở nhiều nơi tại Malaysia, điều này gợi ý rằng sự xuất hiện P. knowlesi ở người có thể đã lan rộng ngoài khu Kapit.
Cox - Sigh và cộng sự đã phát hiện ca sốt rét nhiễm P.knowlesi đầu tiên ở khỉ Macaque đuôi dài (tên khoa học là Macaca fascicularis) khi nhập khẩu từ Singapore đến Ấn Độ vào năm 1931. Một năm sau, Knowles và Das Gupta đã thành công trong việc gây nhiễm thực nghiệm truyền P.knowlesi từ khỉ sang người. Ca bệnh nhiễm P.knowlesi ở người được mô tả đầu tiên vào năm 1965 tại Mỹ, trên một người lính trở về từ Malaysia. Theo phân loại, P. knowlesi thuộc giới Protista, ngành Apicomplexa, lớp Aconoidasida, bộ Haemosporida, họ Plasmodiidae, giống Plasmodium, loài P. knowlesi. Đến năm 1960, tác giả Eyles đã thành công trong việc truyền tác nhân gây bệnh sốt rét ở khỉ là P.cynomolgi cho người trên thực nghiệm. Năm 1966 bằng cách cho muỗi đốt máu có nhiễm KSTSR rồi cho đốt người, tác giả Granharm cũng đã chứng minh rằng các loài KSTSR ở khỉ như P. iuni,, P. knowlesi đều có khả năng truyền bệnh cho người.
Đến năm 1971, chỉ có 2 trường hợp nhiễm P.knowlesi ở người được báo cáo (Chin, 1965 và Yap, 1971), cả 2 ca đều sống ở Peninsular, Malaysia. Sau đó Cox - Sigh và công sự đã tiến hành một nghiên cứu kéo dài từ tháng 3.2000 đến tháng 11.2002 tại Malaysia, Kết quả cho thấy rằng trong số 208 ca bệnh được chẩn đoán ban đầu là nhiễm P.malariae thì có đến 120 ca được xác định là P.knowlesi bằng kỹ thuật PCR lồng (nested PCR). Nhiều trường hợp nhiễm P. knowlesi sau đó cũng được phát hiện tại Kanip, Sarawak, Malaysia năm 2004. Vào năm này một ca bệnh do loài KSTSR này gây nên cũng được phát hiện trên một bệnh nhân làm việc trong rừng ở biên giới giữa Thái và Myanmar (Jongwutiwes và cs., 2000). Một nghiên cứu khác cũng cho hay người bệnh nhiễm P.knowlesi cũng được phát hiện tại Philippines.
Về hình thái học, các nhà nghiên cứu đã quan sát các giai đoạn hồng cầu nhiễm KSTSR được nhuộm Giemsa từ các bệnh nhân chỉ nhiễm đơn thuần P. knowlesixác định bằng phương pháp nested PCR. Các thể tư dưỡng non xuất hiện các thể nhẫn và không thể hoặc rất khó phân biệt với các thể tư dưỡng non của P. falciparum. Có khi xuất hiện nhiều hơn 1 thể nhẫn trong mỗi hồng cầu và hoặc cũng nhìn thấy các chấm hematin đứng đôi. Thể tư dưỡng già có mặt ít hơn 1/3 số hồng cầu bị nhiễm và bào tương đậm đặc, không biến hình, liên quan và dễ nghĩ đến P. malariae. Trong một số trường hợp, chúng ta có thể tìm thấy thể tư dưỡng già dạng “các thể dãi băng” điển hình tương tự với P. malariae. Các thể phân liệt có một cụm trung tâm từ 8 đến 18 tiểu thể hoa cúc và các thể phân liệt già đã không chiếm hết toàn bộ hồng cầu. Các thể tư dưỡng và thể phân liệt được nhuộm sắc tố đậm đặc với sắc tố sốt rét nâu, đậm hoặc màu đen. Các thể giao bào tương tự như giao bào của P. malariae và có hình tròn, chúng chiếm hầu hết thể tích hồng cầu và có sắc tố sốt rét phân tán rải rác. Các hồng cầu nhiễm thể tư dưỡng già và thể phân liệt thường được nhìn thấy trong lam máu giọt đặc, chúng không trương phình và chúng ta không thấy bất cứ chấm đậm và sáng nào; các đặc điểm trên tuy riêng nhưng đôi khi tương tự điển hình đối với các hồng cầu nhiễm P.malariae.
Hình ảnh P. knowlesi soi kính hiển vi của các bệnh nhân
Nhóm nghiên cứu kháng thuốc và nghiên cứu các phương pháp chẩn đoán tại cộng đồng của Khoa Nghiên cứu bệnh sốt rét, Viện Sốt rét - KST - CT Quy Nhơn trong thời gian từ năm 2007 - 2010 khi thực hiện nghiên cứu tại các điểm sốt rét lưu hành của tỉnh Ninh Thuận, Quảng Trị, Gia Lai,…đã nhiều lần phát hiện hình thể ký sinh trùng sốt rét “na ná” như loại P. malariae, nhưng không điển hình như hình ảnh của P.malariae. Tuy nhiên, kết quả này chỉ được bàn luận giữa các kỹ thuật viên có kinh nghiệm của nhóm nghiên cứu về phương diện hình thái học trên kính hiển vi và đặc biệt các trường hợp này đều đáp ứng với thuốc chloroquine và primaquine. Do đó, liệu chăng các trường hợp như thế là P. knowlesi, điều cần thiết là phải xác định ca nghi ngờ bằng kỹ thuật sinh học phân tử, chẳng hạn PCR.
Dịch tễ học nhiễm Plasmodium knowlesi ở Malaysia
Năm 1931, Plasmodium knowlesi lần đầu tiên được biết nhiễm trùng gây tử vong trên các con khỉ đuôi dài. Năm 1932, một mô hình gây nhiễm thực nghiệm sử dụng máu người cho thấy kyus sinh trùng này cũng có thể gây nhiễm trên người. Từ đầu những năm 1930, P. knowlesi đã được sử dụng để điều trị sốt trên những bệnh nhân bị giang mai thần kinh.
Sự truyền bệnh sốt rét từ loài khỉ đến người và người đến người qua trung gian muỗi có thể xảy ra trong điều kiện thực nghiệm. Liệu rằng có một ổ nhiễm lớn về P. knowlesi ở vùng Kapit, đảo Borneo, Malaysia có thể góp phần lớn sự lây truyền từ người đến người hoặc từ khỉ đến người qua muỗi trung gian truyền bệnh thì chưa được rõ thấu đáo. Nhóm muỗi An.leucosphyrus có khả năng lây truyền ký sinh trùng P. knowlesi và có mặt ở vùng Kapit, vì là vật chủ tự nhiên của P. knowlesi, các loài khỉ đuôi dài (Macaca fascicularis) và khỉ có đuôi lợn (Macaca nemestrina). Mặc dầu, hai loài khỉ này sống rất gần gũi con người ở vùng Kapit và là một nguồn tiềm năng mang KSTSR P. knowlesi, nhưng các nhà nghiên cứu vẫn chưa xác định liệu rằng chúng bị nhiễm P. knowlesi. Họ cũng chưa xác định được muỗi truyền bệnh. Các dữ liệu rằng là nhiễm P. knowlesi và những bệnh nhân được phát hiện giống như P. malariae dưới kính hiển vi ở vùng Kapit hầu như là người lớn và rằng là không có các trường hợp mắc bệnh tập trung trong cùng cộng đồng ở chung những căn nhà dài đã gợi ý rằng sự truyền bệnh sự lây truyền xảy ra xa vùng lân cận của những căn nhà dài chung và rằng là sự truyền bệnh đang xảy ra là từ khỉ đến người hơn là từ người qua người. Tuy nhiên, dữ liệu dịch tễ học chỉ được báo cáo các trường hợp bệnh có biểu hiện triệu chứng ở những người tiếp cận các dịch vụ y tế tại các phòng khám và bệnh viện. Nghiên cứu về dịch tễ học sinh học phân tử với qui mô lớn, có thể phát hiện những người nhiễm không triệu chứng, sẽ làm rõ hơn nữa về khả năng người trở thành ổ chứa của P. knowlesi. Họ đã từng nghiên cứu để nhận ra nơi lưu giữ KSTSR loại P. knowlesi trong tự nhiên, muỗi và chu kỳ lây truyền của P. knowlesi ở vùng Kapit.
Người nhiễm tự nhiên với KSTSR loài khỉ, P. knowlesi, cho hơn một nữa số ca bệnh trong nghiên cứu Janet Cox-Singh. Liệu rằng loài KSTSR này đã từng chuyển vật chủ với sự lây truyền bệnh giữa người - người hoặc liệu rằng sự lây truyền giữa loài khỉ qua người cần được thiết lập để có các biện pháp phòng ngừa và kiểm soát thích hợp có thể được thực hiện ở vùng Kapit. Việc phân tích DNA cho P. knowlesi trong số các mẫu máu được phân lập tại vùng này gợi ý rằng phân tích chuổi gen sẽ là hữu ích trong việc làm rõ về số liệu dịch tễ học và tiến triển lâm sàng bệnh sốt rét do P. knowlesi ở người. Nhiễm trùng loài KSTSR P. knowlesi thường chỉ được xem là một loại ký sinh tùng đặc thù cho khỉ đuôi dài (Macaca fascicularis) và khỉ đuôi heo (Macaca nemestrina) nhưng con người lại thường làm việc trong các vùng rừng và hiển nhiên có nguy cơ nhiễm bệnh. Với sự gia tăng tàn phá rừng và sự phát triển của các quốc gia Đông Nam Á, nhiều khỉ trở nên tiếp xúc gần gũi với con người. Vì thế ngày càng có nhiều người sống tại các khu vực ngoại ô bị nhiễm sốt rét loại P. Knowlesi. Loại ký sinh tùng này tìm thấy hầu hết ở các quốc gia Đông Nam Á, đặc biệt ở Borneo, Malaysia, Myanmar, Philippines, Singapore, Thái Lan và các quốc gia láng giềng, nay cũng đã xuất hiện tại Việt Nam. Chúng dường như xảy ra tại các vùng mà trước đây báo cáo không có 4 loại KSTSR cổ điển, muỗi nhiễm cũng chỉ gặp giới hạn trong một vài vùng rừng, trong khi muỗi không nhiễm điển hình hay gặp ở các vùng đô thị nhưng lan truyền có thể xảy ra do sự đa dạng và nhiều lên của quần thể muỗi trong các vùng đó, đặc biệt là Malaysia, những cũng có vài báo cáo tại khu vực biến giới Thái Lan – Myanmar. 1/5 số ca sốt rét được chẩn đoán tại Sarawak, Malaysian Borneo là do P. knowlesi. Loài Plasmodium knowlesi ít lưu hành hơn ở châu Phi. Điều này có thể do nhiều người dân châu Phi mà chính là dân da đen Tây Phi thiếu kháng nguyên Duffy – một loại protein trên bề mặt hồng cầu giúp bảo vệ phần nào theo cơ chế “bệnh chống bệnh” chăng?
Các sinh vật thuộc giống Plasmodium là các ký sinh trùng eukaryotic bắt buộc, một số biết rõ theo tác nhân sinh bệnh sốt rét ở người. Có 4 loại KSTSR nhiễm và gây sốt rét ở người chính là P. falciparum, P. vivax, P. malariae, P. ovale. Mặc dù, các ký sinh trùng thuộc Plasmodium nhiễm trên nhiều loài vật chủ có xương sống khác nhau (bao gồm loài khỉ,, loài gặm nhấm, động vật có móng vuốt, thằn lằn, chim), chúng hiếm khi gây bệnh nghiêm trọng trên các động vật có xương sống, ngoại trừ con người. Loài KSTSR có độc lực nhất đối với con người là P. falciparum và P. vivax (White và cs., 2003), gây ra 300 - 500 triệu ca sốt rét nặng hoặc tử vong trên toàn cầu.
Hiện nay, với sự thông tin có được các nhà khoa học đã hệ thống hóa các loài KSTSR đã thay đổi đáng kể quan 20 năm qua từ các công trình nghiên cứu khoa học chính thống (Perkins và Schall., 2002; Martinsen và cs., 2008) chỉ ra rằng giống Plasmodium không thể từ một nguồn gốc hay từ một tổ tiên (monophyletic) và bao gồm các ký sinh trùng của các giống khác, gồm cả Hepatocystis.
Một số nét đặc trưng của tiến hóa liên quan đến cội nguồn Plasmodium spp
Các vi sinh vật thuộc giống Plasmodium được xác định và phân biệt với các Apicomplexa khác và các sinh vật khác đôi khi nhầm lẫn với KSTSR (Peréz-Tris và cs., 2005), vì KSTSR thực hiện một chu kỳ phát triển gọi là “merogony” (gồm nhiều giai đoạn phân chia nhân theo sau sự phân cắt bào tương sinh ra các tế bào con gọi là merozoites) bên trong hồng cầu và sinh ra các thành phần hạt sắc tố (hemozoin pigment) và các tinh thể là sản phẩm của sự tiêu hóa Hb. Các thành viên khác thuộc bộ Haemosporida biến đổi khác nhau trong việc phối hợp các đặc tính này. Họ Haemoproteidae sinh ra các hạt sắc tố (pigment), nhưng sự phát triển đoạn trứng (merogony) xảy ra trong các mô hơn là trong hồng cầu. Các họ Garniidae và Leucocytozoidae không sinh ra các hạt pigment ở bất kỳ giai đoạn nào, nhưng trường hợp khác có thể thấy phát triển giai đoạn trứng ở các hồng cầu hay tế bào máu.
Tất cả ký sinh trùng sốt rét có một giai đoạn hữu tính xảy ra trong côn trùng ăn máu (blood-feeding insect), là các vật chủ chính đối với các vi sinh vật này (có thể gọi là “vector” về mặt dịch tễ học). Về kinh điển, các loài Plasmodium được mô tả dựa trên đặc điểm hình thái học và đặc điểm tắc lượng hình thái (morphological and morphometric characteristics), đầu tiên giai đoạn trong máu của chu kỳ sinh học. các đặc tính khác sử dụng trước đây để phân loại mức độ độc lực và thời gian chu kỳ hồng cầu (24, 48, 72 giờ).
Đặc điểm về hình thái học thường tỏ ra không đủ để chứng minh và phân biệt các loài và cho thấy trái ngược với các dữ liệu về trình tự phân tử. Các nhà khoa học bắt đầu sử dụng các công cụ và phương pháp tiến bộ để phân loại KSTSR và xây dựng phả hệ di truyền theo một mối quan hệ phả hệ di truyền trong tiến hóa loài.
Trung gian truyền bệnh các loài ký sinh trùng sốt rét có khả năng truyền bệnh từ động vật sang người
Vấn đề bỏ ngõ hiện nay là các nghiên cứu trên thế giới hình như chưa tập trung vào các vật chủ và trung gian truyền bệnh của các loài ký sinh trùng tiềm năng truyền từ động vật sang người. Riêng đối với loài ký sinh tùng ở khỉ nay chuyển sang người cũng là nghiên cứu đi sau. Ban đầu là nghiên cứu của Wharton và Eyles (1961) cho biết trung gian truyền bệnh P. knowlesi từ khỉ cho người là An. hackeri (thuộc nhóm Luecosphyrus), nhưng sau đó người ta khám phá ra rằng An. hackeri không có ái tính với người và chủ yếu truyền bệnh sốt rét cho khỉ (Reid và Weitz., 1961). Một nghiên cứu tiếp theo của Wharton và cộng sự (1962) cho thấy An. latens (nhóm Leucosphyrus) chính là vector truyền bệnh chính loài KSTSR P. inui cho khỉ và P. knowlesi cho cả khỉ và người. An. latens có tập tính đốt người khi chạng vạng cho đến gần sáng và đỉnh điểm là nữa đêm. Loài muỗi này hoạt động chủ yếu trong rừng và bìa rừng nhưng có xu hướng vào nơi ở của người sống bìa rừng và trong rừng để tìm mồi đốt máu (I.Vithylingam., 2005). Kết quả nghiên cứu này cũng cho thấy rằng tỷ lệ nhiễm thoi trùng của An. latens là 0.7% ở bìa rừng và 1.4% trong rừng và chỉ số EIR (Entomological inoculation rate) là 11.98 ở bìa rừng và 14.10 ở trong rừng. Về phân loại khoa học của trung gian truyền bệnh – muỗi An.latens do các tác giả cũng đã được ghi nhận và công nhận như sau: An.latens [Sallum và Peyton (Zulueta 1956, White 1983, Wilkerson 2005)] thì An.latens thuộc giới Animalia, ngành Arthropoda, ngành phụ Hexapoda, lớp Insecta, lớp phụ Pterygota, bộ Diptera, bộ phụ Nematocera, họ Culicidae, loài An. Latens.
Vấn đề bỏ ngõ thứ hai cũng còn rất ít tài liệu đề cập đến sự phân bố cũng như một khía cạnh khác của đặc điểm dịch tễ học của các loài ký sinh trùng sốt rét này, mặc dù trước đây có nghiên cứu trong một thời gian.
Vật chủ và phân bố địa lý một số ký sinh trùng sốt rét đã được nghiên cứu trên thế giới
Loài ký sinh trùng | Vật chủ | Phân bố địa lý |
Plasmodium reichenowi | Tinh tinh | Châu Phi |
Plasmodium falciparum | Con người | Châu Phi, châu Á, Nam và Trung Mỹ |
Plasmodium fieldi | Khỉ đuôi ngắn, giống macaca | Đông Nam Á |
Plasmodium simiovale | Khỉ đuôi ngắn, giống macaca | Đông Nam Á |
Plasmodium hylobati | Khỉ đuôi ngắn, giống macaca | Đông Nam Á |
Plasmodium inui | Khỉ đuôi ngắn, giống macaca | Đông Nam Á |
Plasmodium knowlesi | Khỉ đuôi ngắn, giống macaca | Đông Nam Á |
Plasmodium coatneyi | Khỉ đuôi ngắn, giống macaca | Đông Nam Á |
Plasmodium simium | Khỉ / Nhện | Đông Nam Á |
Plasmodium vivax | Con người | Châu Phi, Á, Nam và Trung Mỹ |
Plasmodium cynomolgi | Khỉ đuôi ngắn, giống macaca | Đông Nam Á |
Plasmodium gonderi | Madrill | Châu Phi |
Plasmodium malariae | Con người | Châu Phi, Á, Nam và Trung Mỹ |
Plasmodium brasilianum | Nhện/Khỉ rú/ /Khỉ đêm | Nam Mỹ |
Plasmodium ovale | Con người | Châu Phi |
Hepatocystis sp. | Dơi/ khỉ | Châu Phi, châu Á |
Plasmodium atheruri | Gặm nhấm | Châu Phi |
Plasmodium vinkei | Gặm nhấm | Châu Phi |
Plasmodium chabaudi | Gặm nhấm | Châu Phi |
Plasmodium berghei | Gặm nhấm | Châu Phi |
Plasmodium yoelii | Gặm nhấm | Châu Phi |
Plasmodium mexicanum | Thằn lằn | Bắc Mỹ |
Plasmodium chiricahuae | Thằn lằn | Bắc Mỹ |
Plasmodium elongatum | Chim | Toàn cầu |
Plasmodium gallinaceum | Chim | Đông Nam Á |
Plasmodium relictum | Chim | Toàn cầu |
Plasmodium floridense | Thằn lằn | Vùng Carribbe / Trung Mỹ |
Plasmodium azurophilum | Thằn lằn | Vùng Carribbe / Trung Mỹ |
Plasmodium faichildi | Thằn lằn | Trung Mỹ |
Plasmodium agamae | Thằn lằn | Châu Phi |
Plasmodium gigantum | Thằn lằn | Châu Phi |
Plasmodium mackerassae | Thằn lằn | Úc |
Chu kỳ sinh học của các Plasmodium spp
KSTSR Plasmodium có chu kỳ phát triển phức tạp, nhiều giai đoạn khác nhau:
1.Giai đoạn nhiễm, khi KSTSR đi vào vật chủ có xương sống thông qua một vector. Giai đoạn này gọi là thoi trùng (sporozoite).
2.Giai đoạn ngoại hồng cầu, trong đó các sporozoite thực hiện nhiều vòng phân chia vô tính (merogony hoặc schizongony) và trưởng thành đến merozoites.
3.Giai đoạn trong hồng cầu, các đơn bào này đi vào các hồng cầu (nhưmerozoites), chuyển dạng thành các thể thực bào (trophozoites) và rồi chúng phân chia thể vô tính thành nhiều merozoites mới (schizont). Trong suốt quá trình schizont, một số KSTSR biệt hóa thành thể sinh sản (gametocytes) hơn là các merozoites xâm nhập. Các giao bào được phân loại thành microgametocytes (sẽ thành gametes đực sau này) và macrogametocytes (sẽ thành gametes sau này). Các giao bào phải trưởng thành thông qua 5 giai đoạn trước khi thành giai đoạn nhiễm để nhiễm vào muỗi.
4.Các giai đoạn sinh sản bắt đầu khi muỗi hút máu từ các vật chủ có nhiễm giao bào trưởng thành. Trong muỗi, các giao bào chuyển dạng thành gamete đực và cái và phát triển thành một zygote. Các zygote này trở thành một ookinate xâm nhập vào các mô của ruột muỗi và cuois cùng thành oocyst. Khi oocyst vở ra hàng ngàn sporozoites lồi ra và chu du trong tuyến nước bọt của muỗi và từ đó chúng sẽ có điều kiện để truyền sang vật chủ khác khi muỗi đốt máu.
Thảo luận về mối quan hệ trong hệ thống phát sinh loài hay phả hệ
Lĩnh vực nghiên cứu về phả hệ di truyền của Plasmodium dương như là một khía cạnh rất được các nhà khoa học quan tâm trong thập niên qua và tiếp tục có nhiều loài hơn nữa được bổ sung vào cây phả hệ và các giả thuyết cũ cũng là vấn đề cần làm rõ trong tương lai đến. Nghiên cứu về phả hệ di truyền cấp độ phân tử của KSTSR bắt đầu vào năm 1991 (Waters và cs., 1991) và hiện nay vẫn đang tiếp tục phát triển. Các kết luận chưa thấu đáo của các nghiên cứu trước đây đã giải thích và làm rõ bởi các nghiên cứu sau này, một mặt vì các hạn chế trước đây trong nghiên cứu là khó khăn trong lựa chọn sự xếp nhóm hay phân loại đầy đủ cũng như lựa chọn gen để phân tích. Việc lựa chọn cả phân loại các vi sinh vật quan tâm (ingroup taxa) và vi sinh vật chưa quan tâm (outgroup taxa) để phân tích phả hệ di truyền có thể có ảnh hưởng có ý nghĩa quan trọng cho các giả thuyết về tiến hóa sinh vật.
Các phân tích phả hệ di truyền cũ thường chứa một số lượng nhỏ các loài trong phân loại cùng nhóm (ingroup), từ 6-12 phân loại có liên quan rất mật thiết nhưng cũng có một số lại khác nhau, chưa thống nhất. Sự phân loại khác nhau như thế chưa rõ ràng dẫn đến khác biệt lớn cũng như đa dạng về giống, phân tích nhiều loài có quan hệ rất xa như “outgroup”, có thể dẫn đến một số kết quả không xác thực và kết luận mâu thuẫn với những gì đã ghi nhận trong y văn thời điểm đó.
Ngoài vấn đề phân loại mẫu, nhiều năm qua nghiên cứu phân loại học về sốt rét (malaria systematics) cũng là một quan tâm lớn bởi các vấn đề vốn có di truyền trong các locus chọn để phân tích. Các small subunit (SSU) rRNA và các protein kết cụm (circumsporozoite protein_CSP) như dữ liệu đáng tin cậy của phả hệ di truyền Plasmodium (Waters và cs., 1991; Waters và cs.,1993; Escalate và Ayala., 1994; Escalante và cs., 1995; McCutchan và cs., 1996; Qari và cs., 1996; Escalante và cs., 1996; Escalante và cs.,1997). Tuy nhiên, các nghiên cứu gần đây chỉ ra không có locuss nào trong số này phù hợp cho vấn đề nghiên cứu tiến hóa củacác loài Plasmodium. SSU rRNA và protein kết cụmnhư là locus chuẩn sử dụng trong hệ thống phân loại mức phân tử cấp độ cao, nhưng sau đó tìm thấy các loài Plasmodium sở hữu các gen riêng biệt của rRNA, mỗi trình diện mỗi điểm khác trong chu kỳ và có thông tin trao đổi di truyền theo các cách khác nhau (Corredor và Enea., 1993). Một số phả hệ di truyền cũ bao gồm một tập hợp paralogs (các gen bản sao) và orthologs (các đồng đẳng thích hợp) và chỉ có orthologs là cây gen đáng tin cậy. Mặc dù các đoạn mồi đặc hiệu cho từng giai đoạn đời sống đã được phát triển cho SSU rRNA loci trong Plasmodium, như sự chuyển dạng gen trong số các paralogs chưa đồng nhất không thể loại trừ, các locus này nên dử dụng thật cẩn thận. Ngoài ra, ngay cả với các phả hệ di truyền gần đây (Leclerc và cs., 2004) sử dụng các trình tự gen cho thấy kết quả không thể giải thích một cách thuyết phục với những ổ gen khác trong bộ gen.
Tương tư như vậy, locus CSP, thường được dùng cho nghiên cứu tiến hóa, có thể khó giải quyết như là mã di truyền đối với protein bề mặt và chịu áp lực lựa chọn mạnh từ hệ thống miễn dịch của các động vật có xương sống. Nhu cầu lựa chọn dưới áp lực, các thay đổi không trung hòatrong gen có thể hoặc là không rõ ràng trong tín hiệu phả hệ di truyền hoặc là dẫn đến kết luận đưa ra các hàm ý không đúng về phả hệ di truyền(Hughes và Hughes., 1995). CSP là một locus ưu thế để nghiên cứu như một số lượng lớn về dữ liệu di truyền sẵn có được quan tâm trong lĩnh vực khoa học sốt rét và có thể xem đây là một nền tảng đặc biệt để chế ra vaccine trong tương lai. Các phương pháp cải tiến cho giải trình tự gen cho phép các loci tiến triển tốt hơn trong quá trình nghiên cứu tiến hóa và CSP giờ đây sử dụng một cách hệ thống.
Vào lúc này, một locus mới được phát triển cho một nghiên cứu phân tử, như là các gen ty thể đối với cytochrome b (Escalante và cộng sự., 1998; Perkins và Schall., 2002) và gen cho enzyme adenylosuccinate (Kedseierki và cộng sự., 2002). Một lời cảnh báo rằng mitochondrial cytochrome b là đích của một số thuốc sốt rét và sự đột biến trong gen được biết có liên quan đến kháng (Vaidya và cs., 1993). Các nghiên cứu này các các nghiên cứu trước đây phù hợp cho các gen đã chọn với hy vọng nghiên cứu phả hệ cho loài Plasmodium dựa trên dữ liệu nhiều locus. Tuy nhiên, nên chú ý rằng cơ hội để xác định các locus liên quan trong loài Plasmodium là đầy hứa hẹn nhờ vào khả năng áp lựa chọn lựa chưa nhận ra từ hệ miễn dịch của vật chủ hoặc (trong trường hợp ký sinh trùng ở người) thuốc, do đó biện pháp hỗ trợ đang được quan tâm.
Một giả định thuộc về trực giác rằng các nghiên cứu về mặt phân tử là một thử thách sớm thật lý tưởng và có liên quan mật thiết với các ký sinh trùng sốt rét ở người. Giờ đây, các công trình nghiên cứu mới đây chỉ ra, mặc dù các loài ký sinh trùng trên loài linh trưởng (ngoại trừ P. falciparum và P. reichenowi) tạo nhóm với nhau, thì các ký sinh tùng sốt rét này với vật chủ người là không liên quan trong nhóm này.
Các mối liên hệ tương đối xa trong số các tác nhân sinh bệnh ở người chỉ ra rằng sốt rét hầu như có liên quan rất xa xưa với các loài linh trưởng (Escalante và cs., 1998; Perkins và Schall, 2002; Leclerc và cs., 2004, Cornejo và Escalante., 2006). Một trong những vấn đề hấp dẫn nhất trong quá trình tiến hóa sốt rét là nguồn gốc bí mật của P. falciparum, một số vấn đề khác đã nghĩ và tổng kết lại trong thời gian 15 năm qua. Các cuốn sách giáo khoa cũ và thậm chí các cuốn sách gần đây đề cập đến P. falciparum như là loài đang tăng truyền bệnh theo chiểu ngang từ chim và đó là sự chuyển dịch vật chủ mà trong đó loài có độc lực P. falciparum thường thấy (White và cs., 2003). Song, các nghiên cứu mới đây hơn bác bỏ lập luận của giả thuyết này, vì P. falciparum rơi vào nhóm động vật có một tổ tiên chung của các ký sinh trùng của động vật có vú, khác và nằm ngoài nhóm đang tác động lên chim và thằn lằn. Mặt khác, các loài cũng nằm bên ngoài của bệnh sốt rét ở loài gặm nhấm và linh trưởng. Đúng hơn là P. falciparum là một nhánh rẽ trong nguồn gốc tổ tiên bao gồm chỉ chính nó và các loài chị em với nó, ký sinh trùng trên loài tinh tinh P. reichenowi.
Giả thuyết nguồn gốc từ chim (avian-origin) xuất phát từ một số nghiên cứu sớm về phả hệ phân tử (molecular phylogenies) cho biết vấn đề do quá trình phân loại học và chọn lọc gen chưa đày đủ (Waters và cs., 1991; Waters và cs., 1993; Escalante và cs., 1995; Escalante và cs., 1996; Escalante và cs., 1997). Lần đầu tiên năm 1996 (Qari và cs., 1996) và tiếp đó làm lại năm 2002 (Perkins và Schall, 2002), bằng chứng mới bác bỏ giả thuyết ngồn gốc từ chim. Perkins và Schall (2002) ấn bản một cây phả hệ tương đối mạnh mẽ và chính xác dựa trên Cytochrome b với một kích cỡ mẫu lớn của ký sinh trùng sốt rét trên động vật có xương sống và một nằm ngoài nhóm của họ chị em. Cây phả hệ Cytochrome b này thay thế P. falciparum trong một đơn vị huyết thống đơn tố của các ký sinh trùng của động vật có vú.
Ngoài ra, đứng về góc độ của ngành nghiên cứu phát sinh chủng loài / hoặc phả hệ di truyền thì phân tích này bao một một thử nghiệm rõ ràng của giả thuyết bắt nguồn từ chim sử dụng thử nghiệm Shimodaira-Hasegawa, một phương pháp dựa trên cây phả hệ tính toán khả năng thay đổi trong phả hệ di truyền này.
Vào lúc này, bằng chứng (Qari và cs., 1996; Escalate và Ayala., 1997; Perkins và Schall., 2002; Leclerc và cs., 2004; Martinsen và cs., 2008) chỉ ra rằng chỉ những loài có quan hệ gần với P. falciparum là P. reichenowi và sự khác biệt của hai loài cách 5 và 8 triệu năm dựa trên thời kỳ hóa thạch của tinh tinh và người (Escalante và Ayala., 1994; Escalante và cs., 1995). Phân tích phả di truyền trong tương lai bao gồm các ký sinh trùng nhiễm vào các con dơi (một số giống KSTSR đã được mô tả là có vật chủ duy nhất) và các động vật có guốc (ungulates) có thể sẽ giúp xác định các mối quan hệ gần của P. falciparum và hiểu biết hơn về lịch sử tiến hóa của các ký sinh trùng này.
Một số hình ảnh minh họa
KSTSR Plasmodium các giai đoạn trong hồng cầu:
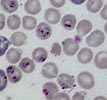
| Tên khoa học: Giới: G.đoạn trong chu kỳ: | Plasmodium chabaudi Vô tính Thể nhẫn và thể tư dưỡng trẻ | |
Plasmodium chabaudi | Nguồn | PHIL_2725_lores.jpg?w=900 | |
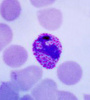
| Tên khoa học: Giới: G.đoạn trong chu kỳ: | Plasmodium vivax Vô tính Tư dưỡng trưởng thành | |
Plasmodium vivax | Nguồn | PHIL_2725_lores.jpg?w=900 | |
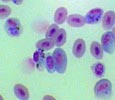
| Tên khoa học: Giới: | Plasmodium cathemerium Vô tính |
| | | | |
Tài liệu tham khảo
1.Coatney GR, Collins WE, Warren M, Contacos PG. 1971. The Primate Malarias. US Department of Health, Education and Welfare, Bethesda.
2.Cornejo OE, Escalante AA. 2006. The origin and age of Plasmodium vivax. Trends Parasitol. 22(12):558-63.
3.Corredor V, Enea V. 1993. Plasmodial ribosomal RNA as phylogenetic probe: a cautionary note. Mol. Biol. Evol. 10:924-926.
4.Escalante AA, Ayala FJ. 1994. Phylogeny of the malarial genus Plasmodium, derived from rRNA gene sequences. Proc. Natl. Acad. Sci. USA 91:11373-11377.
5.Escalante AA, Barrio E, Ayala FJ. 1995. Evolutionary origin of human and primate malarias: evidence from the circumsporozoite protein. Mol. Biol. Evol. 12:616-626.
6.Escalante AA, Freeland DE, Collins, WE, Lal AA. 1998. The evolution of primate malaria parasites based on the gene encoding cytochrome b from the linear mitochondrial genome. Proc. Natl. Acad. Sci. USA 95:8124-8129.
7.Escalante AA, Goldman IF, De Rijk P, De Wachter R, Collins WE, Qari SH, Lal. AA 1997. Phylogenetic study of the genus Plasmodium based on the secondary structure-based alignment of the small subunit ribosomal RNA. Mol. Biochem. Parasitol. 90:317-321.
8.Hughes MK, Hughes AL. 1995. Natural selection on Plasmodium surface proteins. Mol Biochem Parasitol. 71(1):99-113.
9.Kedzierski L, Escalante AA, Isea R, Black CG, Barnwell JW, Coppel RL. 2002. Phylogenetic analysis of the genus Plasmodium based on the gene encoding adenylosuccinate lyase. Infect Genet Evol. 1(4):297-301.
10.Leclerc MC, Hugot JP, Durand P, Renaud F. Evolutionary relationships between 15 Plasmodium species from new and old world primates (including humans): an 18S rDNA cladistic analysis. 2004. Parasitology. 129(6):677-84.
11.Martinsen ES, Perkins SL, Schall JJ. 2008. A three-genome phylogeny of malaria parasites (Plasmodium and closely related genera): Evolution of life-history traits and host switches. Mol Phylogenet Evol 47(1):261-273.
12.McCutchan TF, Kissinger JC, Touray MG, Rogers MJ, Li J, Sullivan M, Braga EM, Krettli AU, Miller LH. 1996.Comparison of circumsporozoite proteins from avian and mammalian malarias: biological and phylogenetic implications. Proc Natl Acad Sci U S A. 93:11889-94.
13.Perez-Tris J, Hasselquist D, Hellgren O, Krizanauskiene A, Waldenstrom J, Bensch S. 2005. What are malaria parasites? Trends Parasitol. 21(5):209-11.
14.Perkins SL, Sarkar IN, Carter R. 2007. The phylogeny of rodent malaria parasites: simultaneous analysis across three genomes. Infect Genet Evol. 7(1):74-83.
15.Perkins SL, Schall JJ. 2002. A molecular phylogeny of malarial parasites recovered from cytochrome b gene sequences. J Parasitol. 88(5):972-8.
16.Qari SH, Shi YP, Pieniazek NJ, Collins WE, Lal AA. 1996. Phylogenetic Relationship among the Malaria Parasites Based on Small Subunit rRNA Gene Sequences: Monophyletic Nature of the Human Malaria Parasite, Plasmodium falciparum. Mol. Phylo. Evol. 6(1)157-165.
17.Vaidya AB, Lashgari MS, Pologe LG, Morrisey J. 1993. Structural features of Plasmodium cytochrome b that may underlie susceptibility to 8-aminoquinolines and hydroxynaphthoquinones. Mol Biochem Parasitol. 58(1):33-42.
18.Vargas-Serrato E, Corredor V, Galinski MR. 2003. Phylogenetic analysis of CSP and MSP-9 gene sequences demonstrates the close relationship of Plasmodium coatneyi to Plasmodium knowlesi. Infect. Genet. Evol. 3(1):67-73.
19.Waters AP, Higgins DG, McCutchan TF. 1991. Plasmodium falciparum appears to have arisen as a result of a lateral transfer between avian and human hosts. Proc. Natl. Acad. Sci. USA 88:3140-3144
20.Waters AP, Higgins DG, McCutchan TF. 1993. Evolutionary relatedness of some primate models of Plasmodium. Mol Biol Evol. 10(4):914-23.
21.White NJ. 2003. Malaria, pp. 1205-1296. In G. C. Cook, and A. Zumla. Mansonís Tropical Diseases, 21st ed. WB Saunders, Philadelphia.
22.Bronner U., Divis P.C., Farnert A., Singh B. (2009). Swedish traveller with Plasmodium knowlesi malaria after visiting Malaysian Borneo. Malar J. 8: 15, PMID 19146706, Full text at PMC: 2634766, doi:10.1186/1475-2875-8-15.
23.Chin W, Contacos PG, Collins WE, Jeter MH, Alpert E. Experimental mosquito-transmission of Plasmodium knowlesi to man and monkey. Am J Trop Med Hyg 1968; 17: 355-358. MEDLINE
24.Chin W, Contacos PG, Coatney RG, Kimbal HR. (1965). "A naturally acquired quotidian-type malaria in man transferable to monkeys". Science 149: 865. doi:10.1126/science.149.3686.865. PMID 14332847.
25.Coatney GR, Collins WE, Warren M, Contacos PG. The primate malarias [original book published 1971] [CD-ROM]. Version 1.0. Atlanta: Centers for Disease Control and Prevention; 2003.
26.Cox-Singh J, Davis TM, Lee KS, Shamsul SS, Matusop A, Ratnam S, Rahman HA, Conway DJ, Singh B (2008). "Plasmodium knowlesi malaria in humans is widely distributed and potentially life threatening." Clin. Infect. Dis. 46(2): 165-171, PMID 18171245, Full text at PMC: 2533694, Template: DOI10.1086/524888.
27.Cogswell F.B. (1992). The hypnozoite and relapse in primate malaria. Clin. Microbiol. Rev 5(1): 26-35, PMID 1735093, Full text at PMC: 358221.
28.Cox-Singh J, Hiu J, Lucas SB, Divis PC, Zulkarnaen M, Chandran P, Wong KT, Adem P, Zaki SR, Singh B, Krishna S.(2010) Severe malaria - a case of fatal Plasmodium knowlesi infection with post-mortem findings: a case report. Malar J. 9(1):10
29.Daneshvar C., Davis T.M.E., Cox-Singh J., et al. (2009). "Clinical and Laboratory Features of Human Plasmodium knowlesi Infection." Clin Infect Dis 49(6): 852–860, doi:10.1086/605439
30.Escalante AA, Freeland DE, Collins WE, Lal AA. The evolution of primate malaria parasites based on the gene encoding cytochrome b from the linear mitochondrial genome. Proc Natl Acad Sci U S A. 1998; 95:8124–9..
31.Fong YL, Cadigan FC, Coatney GR. A presumptive case of naturally occurring Plasmodium knowlesi malaria in man in Malaysia. Trans R Soc Trop Med Hyg. 1971;65:839 – 40.
32.MMWR Weekly. Simian malaria in a U.S traveler -New York, 2008. March 13, 2009/58(09):229-232.
33.Mendis K, Sina B, Marchesini P, Carter R (2001). "The neglected burden of Plasmodium vivax malaria.". Am J Trop Med Hyg 64 (1-2 Suppl): 97-106. PMID 11425182.
34.Singh B et al (2004). A large focus of naturally acquired Plasmodium knowlesi infections in human beings. Lancet 2004;363:1017-102.
35.Janet Cox-Singh, Timothy M.E. Davis, Kim-Sung Lee et al (2008). Plasmodium knowlesi Malaria in Humans Is Widely Distributed and Potentially Life Threatening. Clinical Infectious Diseases 2008:46;165-711.
36.Somchai Jonwutiwes, Chaturong Putaporntip, Takuya Iwasaki, Tetsutaro Sata, and Hiroji Kanbara (2004). Naturally acquired Plasmodium knowlesi malaria in human, Thailand. Emerging Infectious Diseases, Vol.10,No.12, November 2004
37.Somchai Jongwutiwes, Chaturong Putaporntip, Takuya Iwasaki, Tetsutaro Sata, and Hiroji Kanbara (2004). Emerging Infectious Diseases. Vol. 10, No. 12, Dec. 2004
38.Waters AP, Higgins DG, McCutchan TF. Evolutionary relatedness of some primate models of Plasmodium. Mol Biol Evol. 1993;10:914–23.
39.White N.J. (2008). Plasmodium knowlesi: The fifth Human Malaria Parasite. CID 2008:46
40.Wolfe ND, Escalante AA, Karesh WB, Kilbourn A, Spielman A, Lal AA. Wild primate populations in emerging infectious disease research: the missing link? Emerg Infect Dis. 1998;4:149–58.
41.Yap FL, Cadigan FC, Coatney GR. (1971). "A presumptive case of naturally occurring Plasmodium knowlesi malaria in man in Malaysia". Trans R Soc Trop Med Hyg 65 (6): 839–40.
42.Singh B, Lee KS, Matusop A, Radhakrishnan A, Shamsul SSG, Cox-Singh J, Thomas A, Conway DJ (2004). "A large focus of naturally acquired Plasmodium knowlesi infections in human beings". Lancet 363: 1017–24.
43.Jongwutiwes S, Putaporntip C, Iwasaki T, Sata T, Kanbara H. (2004). "Naturally acquired Plasmodium knowlesi malaria in human, Thailand". Emerg Infect Dis 10 (12): 2211–3.
44.Vythilingam I, Tan CH, Asmad M, Chan ST, Lee KS, Singh B. (2006). "Natural transmission of Plasmodium knowlesi to humans by Anopheles latens in Sarawak, Malaysia". Trans R Soc Trop Med Hyg 100: 1087–88.
45.Wharton RH, Eyles DE. (1961). "Anopheles hackeri, a vector of Plasmodium knowlesi in Malaysia". Science 134: 279–80.
46."Plasmodium knowlesi Malaria in Humans Is Widely Distributed and Potentially Life Threatening". Clinical Infectious Diseases (University of Chicago Press/Infectious Diseases Society of America) 46: 165—171.
47.Plasmodium knowlesi: The Fifth Human Malaria Parasite – N.J.White, Center for Vaccinologyand Tropical Medicine, Oxford, UK (Editorial Commentary).
48.Is a monkey malaria from Borneo an emerging human disease? Thomas F McCutChan. National Allergy and Infectious Diseases, USA (Editorial).
49.Peter Van de Eede1, Hong Nguyen Van, Chantal Van Overmeir, Indra Vythilingam, Thang Ngo Duc, Hung Le Xuan, Hung Nguyen Manh, Umberto D'alseeandro, Annette Erhart. First report on human P. knowlesi infections in Vietnam.
50.Jongwutiwes S, Putaporntip C, Iwasaki T, Sata T, Kanbara H. (2004). "Naturally acquired Plasmodium knowlesi malaria in human, Thailand". Emerg. Infect. Dis 10 (12): 2211–3. PMID 15663864.
51.McCutchan TF, Piper RC, Makler MT (2008). "Use of malaria rapid diagnostic test to identify Plasmodium knowlesi infection." Emerg Infect Dis 14(11): 1750-1752, PMID 18976561, Full text at PMC: 2630758.
52.Singh B, Lee KS, Matusop A, Radhakrishnan A, Shamsul SSG, Cox-Singh J, Thomas A, Conway DJ (2004). "A large focus of naturally acquired Plasmodium knowlesi infections in human beings". Lancet363 (9414): 1017–24. doi:10.1016/S0140-6736(04)15836-4.
53.Haynes JD, Dalton JP, Klotz FW, McGinniss MH, Hadley TJ, Hudson DE, Miller LH (1988). "Receptor-like specificity of a Plasmodium knowlesi malarial protein that binds to duffy antigen ligands on erythrocytes." J Exp Med 167(6): 1873-1881, PMID 2838562, Full text at PMC: 2189679.
54.Vythilingam I, Noorazian YM, Huat TC, Jiram AI, Yusri YM, Azahari AH, Norparina I, Noorrain A, Lokmanhakim S (2008). "Plasmodium knowlesi in humans, macaques and mosquitoes in peninsular malaysia." Parasit Vectors 1(1): 26, PMID 18710577, Full text at PMC: 2531168, doi:10.1186/1756-3305-1-26.
55.Lee K.S., Cox-Singh J., Brooke G., Matusop A., Singh B (2009). "Plasmodium knowlesi from archival blood films: Further evidence that human infections are widely distributed and not newly emergent in Malaysian Borneo." Int J Parasitol, doi:10.1016/j.ijpara.2009.03.003
56.Manson-Bahr PEC, Bell DR, eds. (1987). Manson's Tropical Diseases. London: Bailliere Tindall, ISBN 0702011878.
57.Ng O.T., Ooi E.E., Lee C.C., Lee P.J., Ng L.C., Pei S.W., Tu T.M., Loh J.P., Leo Y.S. (2008) Naturally acquired human Plasmodium knowlesi infection, Singapore. Emerg. Infect. Dis. 14(5): 814-816, PMID 18439370, Full text at PMC: 2600232
58.Yap FL, Cadigan FC, Coatney GR. (1971). "A presumptive case of naturally occurring Plasmodium knowlesi malaria in man in Malaysia". Trans R Soc Trop Med Hyg 65 (6): 839–40. doi:10.1016/0035-9203(71)90103-9. PMID 5003320.
59.Vythilingam I, Tan CH, Asmad M, Chan ST, Lee KS, Singh B. (2006). "Natural transmission of Plasmodium knowlesi to humans by Anopheles latens in Sarawak, Malaysia". Trans R Soc Trop Med Hyg 100: 1087–88. doi:10.1016/j.trstmh.2006.02.006.